极地中心在极地假交替单胞菌基因编辑体系构建研究中取得新进展
发布日期:2025-11-11 09:00:03
基因编辑技术,特别是以高效、精准和可编程著称的CRISPR/Cas系统,已成为功能基因组学研究的革命性工具,并深刻变革了微生物遗传操作范式。然而,极地野生细菌因其遗传背景不清、基因编辑工具不兼容、密码子偏好差异及遗传元件匮乏等复合挑战,构成了亟待破解的“黑箱”困境,导致其高效遗传操作体系至今尚属空白。这一瓶颈严重制约了我们对极地微生物环境适应机制的深入解析及其基因资源应用潜力的开发。
近日,极地中心极地环境监测中心廖丽研究员课题组在极地海洋细菌Pseudoalteromonas fuliginea中成功建立了基于CRISPR/Cas9的高效基因编辑体系,实现了高成功率的基因敲除与敲入等基因操作,为极地微生物的功能基因组学研究提供了关键技术支撑。相关成果发表于美国微生物学会旗下经典期刊《Applied and Environmental Microbiology》。论文题为 “Development of a CRISPR/Cas9-induced gene editing system for Pseudoalteromonas fuliginea and its applications in functional genomics”,第一作者为中国极地研究中心与上海交通大学联合培养博士生段泽东,唯一通讯作者为廖丽研究员。

该研究基于P. fuliginea的生理特性与遗传背景,设计并构建了一套适用于该菌的CRISPR/Cas9基因编辑系统。通过筛选高效启动子、优化Cas9编码序列及整合sgRNA模块,研究人员成功实现了精准、高效的基因编辑。与传统双交换同源重组方法相比,该体系在多个目标位点上显著提升了编辑成功率,由原先的0.1%~2%提升到70%~90%以上。研究人员利用该体系对多种功能基因进行了编辑验证,包括运动相关基因fliJ、代谢调控基因csrA、色素合成基因indA以及sRNA (Pf1–Pf3)等。其中,对fliJ基因的敲除导致菌株运动能力显著下降,回补后表型恢复至野生型水平(图1);对indA基因的编辑则影响了色素合成,成功验证了体系的准确性与可靠性。此外,该体系还实现了蛋白标签化(如csrA::3×FLAG与tse2::gfp)操作,进一步拓展了其在功能验证与定位研究中的应用。
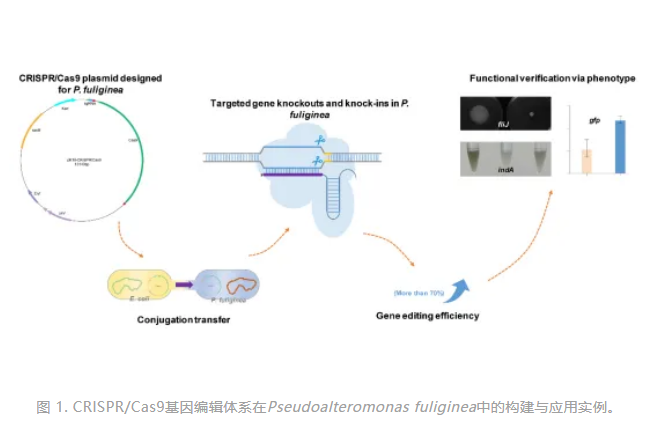
该研究建立的CRISPR/Cas9基因编辑系统,有效解决了极地海洋细菌P. fuliginea在遗传操作上的技术限制,大幅提升了基因功能研究的效率与可操作性。该体系为揭示极地海洋细菌在环境适应、信号调控及宿主互作等过程中的分子机制提供了关键工具,也为极地微生物资源在海洋生物技术与合成生物学领域的应用开发奠定了坚实基础。本研究得到了国家重点研发计划(2022YFC2807501)和国家自然科学基金(42476264、41976224)支持。
课题组围绕极地假交替单胞菌遗传操作体系建立及其环境适应相关分子机制发表的论文:
1. Duan Z, Yang R, Lai T, Jiang W, Zhang J, Chen B, Liao L.* Development of a CRISPR/Cas9-induced gene editing system for Pseudoalteromonas fuliginea and its applications in functional genomics. Appl Environ Microb l0:e01771-25 (2025). https://doi.org/10.1128/aem.01771-25
2. Duan, Z., Liao, L.*, Lai, T. et al. Dynamic and intricate regulation by the Csr sRNAs in the Arctic Pseudoalteromonas fuliginea. Commun Biol 8, 369 (2025). https://doi.org/10.1038/s42003-025-07780-y.
3. Wen J, Liao L*, Duan Z, Su S, Zhang J, Chen B. Identification and regulatory roles of a new Csr small RNA from Arctic Pseudoalteromonas fuliginea BSW20308 in temperature responses. Microbiol Spectr (2023) e0409422. https://doi.org/10.1128/spectrum.04094-22
4. Liao L*, Liu C, Zeng Y, Zhao B, Zhang J, Chen B. Multipartite genomes and the sRNome in response to temperature stress of an Arctic Pseudoalteromonas fuliginea BSW20308. Environ Microbiol (2019) 21:272-285. https://doi.org/10.1111/1462-2920.14455
来源:中国极地研究中心(中国极地研究所)





 沪公网安备3101150201798号
沪公网安备3101150201798号